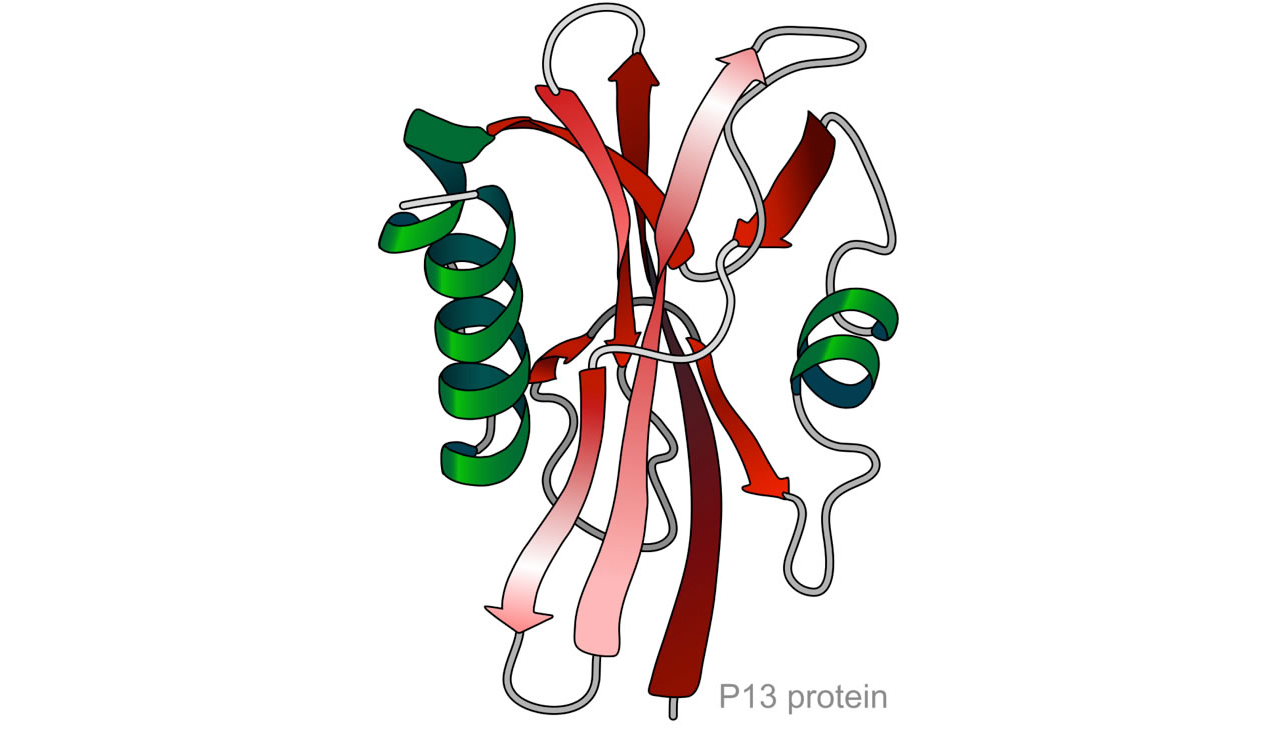
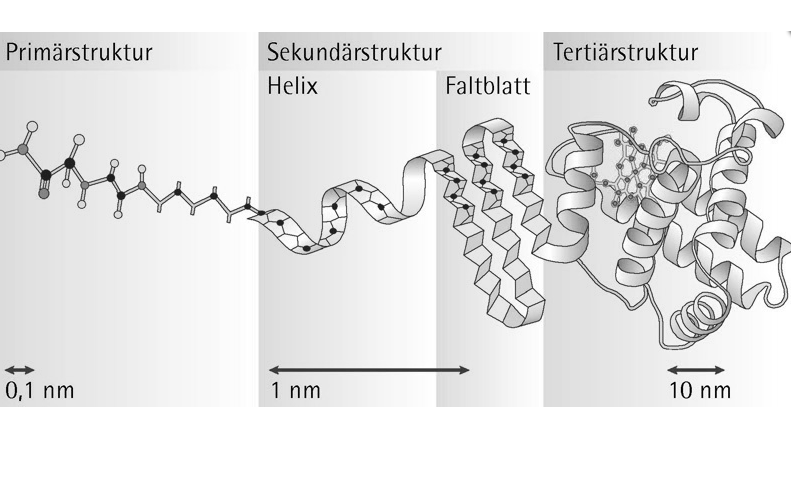
Bändermodelle sind Molekülmodelle von Proteinstrukturen. Sie visualisieren Elemente der Sekundärstruktur wie eine α-Helix oder ein β-Faltblatt und erlauben eine Darstellung der Tertiärstruktur. Verschiedene Polypeptide können zur besseren Unterscheidung farblich unterschiedlich markiert sein. Bändermodelle werden bei der molekularen Modellierung eingesetzt, z. B. beim Protein-Engineering. Das Bändermodell wurde 1980/81 durch Jane Richardson etabliert. Ähnliche Ansätze gab es aber schon früher.
Strukturdarstellungen
Software
Verschiedene Programme zur Darstellung von Proteinen im Bändermodell wurden entwickelt, z. B. Molscript von Arthur M. Lesk, Karl Hardman und John Priestle, Jmol, DeepView, MolMol, KiNG, UCSF Chimera und PyMOL von Warren DeLano.
Literatur
- Peptide und Proteine
Einzelnachweise
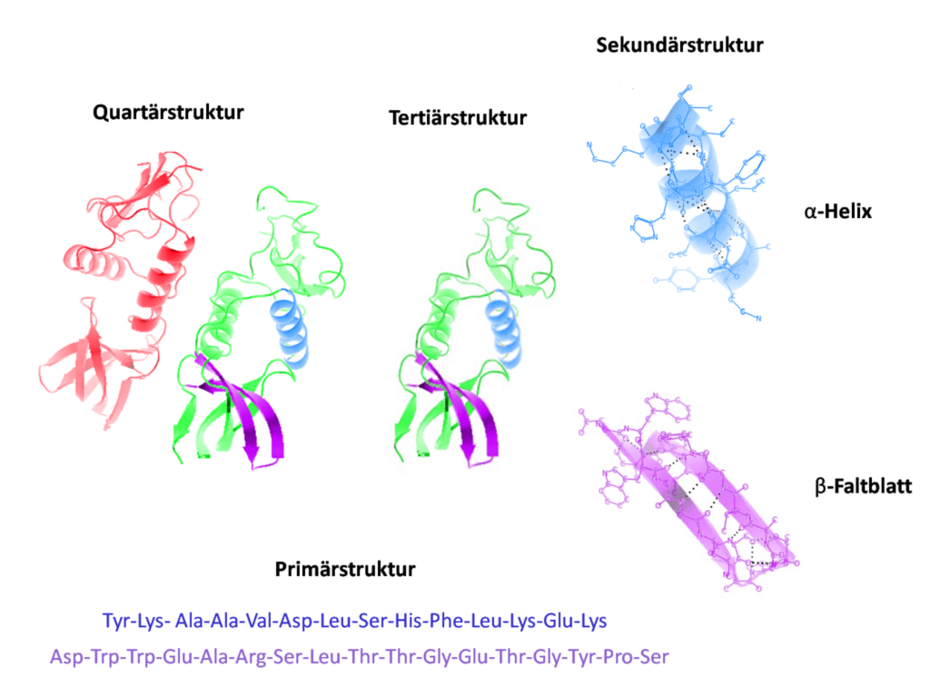
![Proteine • Aufbau und Funktion · [mit Video]](https://d1g9li960vagp7.cloudfront.net/wp-content/uploads/2021/08/WP_Proteine_4-1024x576.jpg)